Interruptor molecular: el nuevo truco de los antibióticos
Mediante el escaneo de compuestos que pueden bloquear rutas de biosíntesis bacterianas, se ha descubierto una alternativa antibiótica que inhibe el crecimiento de patógenos actuando selectivamente sobre un interruptor molecular en una estructura regulatoria del ARN. Estos interruptores, que corresponden a elementos muy importantes para bacterias, se denominan riboswitches o interruptores de ARN, que son capaces de reconocer metabolitos como vitaminas y otras coenzimas, modulando el proceso de traducción de proteínas. Las nuevas alternativas de antibióticos que se unan a estos elementos reguladores del ARN mensajero permitirán alterar los niveles de producción de proteínas relevantes para rutas de biosíntesis de metabolitos que sean esenciales para microorganismos, de forma de frenar su proliferación y controlar enfermedades.
Inhibición de biosíntesis de vitamina B2
La edad de oro del descubrimiento de antibióticos, entre 1940 y 1960, fue anunciada por el trabajo de Selman Waksman (Waksman, S. A. Mycologia 39, 565–569 (1947)), bioquímico y microbiólogo que acuñó el término "antibiótico" y que fue el primero en utilizar un screening sistemático para descubrir efectos antibacterianos. Esfuerzos realizados en su laboratorio produjeron más de 20 antibióticos naturales – el más notable, el caso de estreptomicina en 1943, el primer tratamiento efectivo para la tuberculosis. El descubrimiento llevó a Waksman a ser premiado con el Premio Nobel de Fisiología o Medicina en 1952. Esta investigación llamó la atención de científicos y de farmacéuticas estadounidenses y conllevó a la cooperación para el uso clínico de la estreptomicina. En una publicación reciente (Nature http://dx.doi.org/10.1038/nature15542), John A. Howe y colaboradores, describieron una novedosa estrategia antibacteriana utilizando un sofisticado refinamiento del screening fenotípico introducido por Waksman.
Siete décadas después de la investigación de Waksman, la generación de antibióticos naturales ha disminuido, y por diversas razones, existen pocas investigaciones farmacológicas activas al respecto. Esto es una necesidad clínica urgente, a pesar del aumento de la resistencia de antibióticos existentes. En este contexto, el sorprendente descubrimiento de Howe y su equipo sobre un medicamento que apunta al ARN no codificante bacteriano (ncRNA, por sus siglas en inglés) proporciona un punto de partida brillante.
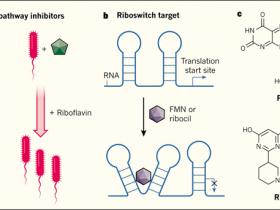
La detección fenotípica implica la aplicación de compuestos a cultivos bacterianos y observar el cambio de las características celulares (de su fenotipo). Desde los días de Waksman, la determinación fenotípica más simple para actividad antibacteriana ha sido buscar la inhibición del crecimiento -la muerte celular es el fenotipo más severo y se mide fácilmente. Howe y colegas establecieron una técnica más sutil que involucra una única vía metabólica bacteriana. A diferencia de los seres humanos, las bacterias pueden sintetizar el metabolito riboflavina, también llamada vitamina B2, que es un precursor de cofactores necesarios en varias reacciones enzimáticas. El más destacado de ellos es el flavín mononucleótido (FMN). Los genes implicados en la biosíntesis de riboflavina son esenciales sólo cuando las bacterias no pueden adquirir la vitamina del entorno. Por esto, los investigadores plantearon la hipótesis que los compuestos antibacterianos, cuyos efectos inhibitorios sobre el crecimiento pueden ser revertidos suplementando riboflavina al medio de cultivo, podrían ser candidatos para la inhibición de la ruta de síntesis de riboflavina (fig. 1a). En esta investigación se probaron alrededor de 57.000 compuestos sintéticos, identificando a la molécula robocil como capaz de eliminar selectivamente bacterias mediante el bloqueo de la biosíntesis de riboflavina.
Para confirmar el mecanismo de acción de ribocil, los autores demostraron que las bacterias tratadas con los compuestos estaban libres de roboflavina. En ratones infectados con bacterias patógenas, el tratamiento con ribocil redujo la concentración de bacterias en más de 1.000 veces, lo cual es un prometedor comienzo. El equipo identificó el blanco molecular del ribocil, aislando clones de bacterias que se volvieron resistentes durante exposiciones prolongadas a esta molécula a concentraciones subletales, posteriormente secuenciaron su genoma. Lo que inicialmente pareció un ejercicio rutinario, se volvió emocionante cuando se encontró que las bacterias resistentes poseían mutaciones en regiones no codificantes del genoma, sugiriendo que robocil bloquea un mecanismo de regulación génica, en lugar de inhibir una proteína como blanco. El sitio de ataque de ribocil, condujo a los investigadores a identificar un dominio en ncRNA altamente estructurado, río arriba de la secuencia que marca el inicio de la traducción en el ARN mensajero codificante para una enzima sintasa clave para la biosíntesis de riboflavina. Resulta que el dominio del ncRNA al que se une ribocil, normalmente está acoplado a FMN, y este sitio corresponde a un riboswitch término utilizado para regiones del ARN que cambian su estructura cuando se unen a un ligando (fig. 1b). La unión de FMN entrampa al riboswitch en una configuración que impide el acceso de la maquinario de transcripción y traducción y por lo tanto, bloquea la expresión de la enzima sintasa. Este mecanismo proporciona una manera para que bacterias reduzcan la producción excesiva de riboflavina, y por consecuencia de FMN cuando ya hay suficiente disponibilidad del cofactor. Al unirse al mismo blanco en el ncRNA, el ribocil engaña al riboswitch de forma que este responde apagando la producción de riboflavina y privando a las bacterias del metabolito. Como una evidencia de peso, los investigadores utilizaron cristalografía de rayos X para proporcionar una imagen instantánea del ribocil uniéndose al riboswitch del transcrito. Esta información estructural será valiosa para mejorar la actividad antibacteriana de los derivados de ribocil para su potencial uso clínico.

Figura 1: el bloqueo de la vía metabólica identifica un antibiótico
a, las bacterias utilizan una vía metabólica para sintetizar riboflavina - una molécula implicada en muchas reacciones enzimáticas - cuando no está disponible en el ambiente. Howe y colegas buscaron inhibidores de esta vía identificando compuestos antibacterianos cuya acción podría atenuarse mediante la adición de riboflavina. b, el screening identificó a la molécula sintética ribocil como un inhibidor selectivo de la biosíntesis de riboflavina. Los autores reportaron que ribocil une a una un riboswitch, dominio regulador de una zona no codificante del ARN mensajero que contiene información para la generación de una enzima sintasa involucrada en la síntesis de riboflavina. El riboswitch está acoplado normalmente a flavín mononucleótido (FMN), metabolito producido a partir de riboflavina; tal unión induce un cambio estructural en el riboswitch que impide la expresión de la enzima sintasa, deteniendo por lo tanto la producción riboflavina cuando hay suficiente disponibilidad de FMN. c, el ribosil, a pesar de tener una estructura muy diferente de la FMN, también se une este sitio, engañando al interruptor, apagando la síntesis de riboflavina y privando a las bacterias de este metabolito esencial.
Este mecanismo de imitación de un ligando natural de un riboswitch no se ha descrito para otros compuestos antibacterianos. Así, el estudio demuestra de manera concluyente las ventajas de la detección fenotípica, la que permite evaluar simultáneamente de una manera imparcial todos los componentes constituyentes de una vía – tanto proteínas como ácidos nucleicos. Aunque los riboswitch son dianas obvias para el descubrimiento de antibacterianos, Howe y colegas han identificado por primera vez una molécula de unión a riboswitch que no es un análogo estructural de un metabolito-ligando. Esto mejora las probabilidades de que ribocil no tendrá efectos fuera de su objetivo alterando otras vías que involucran riboflavina y FMN, lo cual ya fue insinuado por las observaciones de los autores, quienes incluso ensayando altas dosis del compuesto, no presenciaron efectos tóxicos en ratones. Existen sólo unos pocos casos previos, de elementos de unión a ncRNA que han sido explotados como potenciales fármacos; un caso es el de una región en el virus de hepatitis C a la que se unen inhibidores de la síntesis de proteínas virales. Pero muchos antibióticos naturales funcionan interfiriendo con la síntesis de proteínas a través de la unión a componentes de ncRNA en el ribosoma bacteriano, la máquina celular que sintetiza todas las proteínas.
Asumiendo la naturaleza de compuestos que se unen a regiones del ncRNA en el ribosoma, miradas optimistas podrían apuntar a que la gran cantidad de ncRNAs recientemente descubiertos en los genomas bacterianos y en organismos más complejos proporcionará una ampliación de la gama de objetivos para la intervención terapéutica. Para los escépticos, el trabajo de Howe y su equipo podría disipar las dudas que elementos dirigidos a ncRNA puedan ofrecer nuevas opciones de medicamentos. Adicionalmente, se podría esperar que médicos que necesitan urgentemente antibióticos alternativos, estén entre los primeros en aprovechar los beneficios de fármacos emergentes dirigidos al ARN.
Fuente bibliográfica
Non-coding RNA: Antibiotic tricks a switch
Thomas Hermann
Department of Chemistry and Biochemistry, University of California, San Diego, La Jolla, California 92093, USA.
doi:10.1038/nature15635